Wenn Dinge zu klein sind, um sie mit bloßem Auge zu sehen, wie es bei vielen biologischen Systemen der Fall ist, dann verwendet man ein Mikroskop. Viele hochinteressante biologische Strukturen sind jedoch so klein, dass selbst ein Mikroskop sie nicht auflösen kann, zumindest kein Standard-Lichtmikroskop. Das liegt daran, dass Licht selbst eine bestimmte Größe hat. Dinge, die kleiner sind als die Hälfte der Wellenlänge des für die Abbildung verwendeten Lichts können nicht aufgelöst werden, und da die kleinste Wellenlänge des sichtbaren (blauen) Lichts etwa 400 nm beträgt, kann die Standard-Lichtmikroskopie die Details von Strukturen unter 200 nm nicht auflösen. Es wurden jedoch bereits mehrere Techniken entwickelt, um diese Grenze zu umgehen.
Ein Trick besteht darin, einzelne fluoreszierende Moleküle abzubilden, z. B. durch den Einsatz spezieller fluoreszierender Proteine, die auf Befehl zum Blinken gebracht werden können. Jeder einzelne kleine Lichtpunkt, den das Mikroskop erkennt, kann dann nicht besser aufgelöst werden als mit einem normalen Lichtmikroskop. Da wir aber wissen, dass es sich bei der Lichtquelle um ein einzelnes Molekül handelt, lässt sich die genaue Position des Fluorophors mit viel höherer Genauigkeit zurückverfolgen. Diese Technik wird als Einzelmolekül-Lokalisierungsmikroskopie bezeichnet.
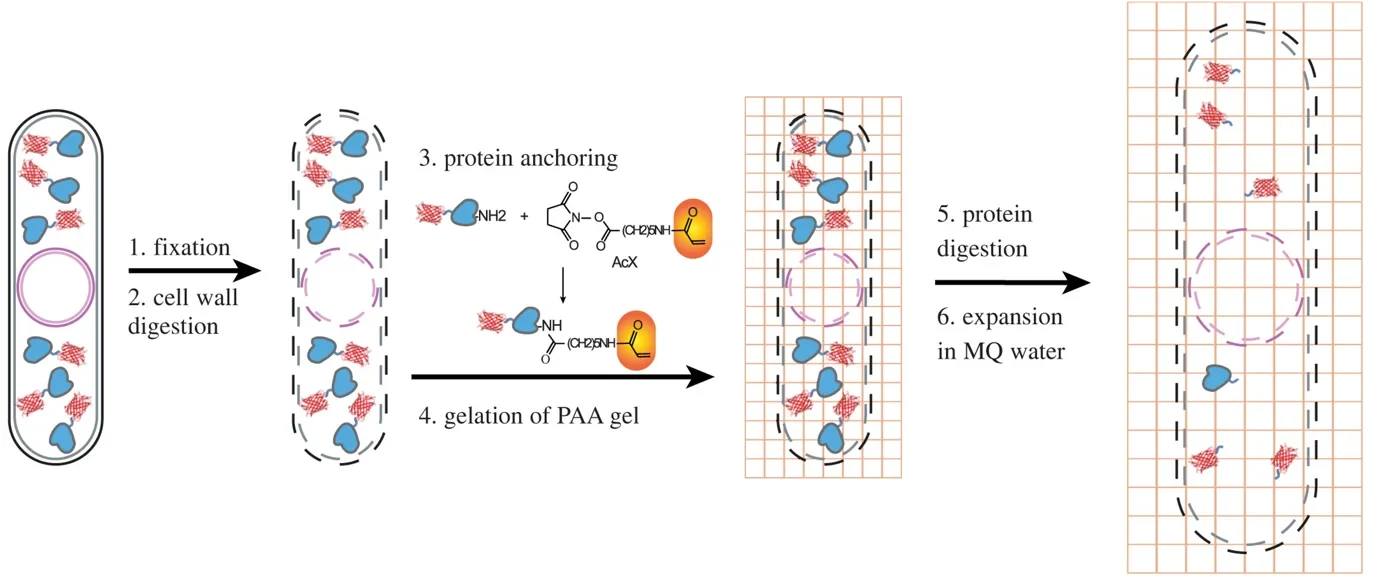
Ein weiterer Trick besteht darin, die Probe selbst zu vergrößern. Auch hier werden fluoreszierende Moleküle in Kombination mit speziellen Kunststoffpolymeren verwendet (die übrigens auch eine wichtige Rolle in Windeln dabei spielen, Babys trocken zu halten). Die Fluorophore werden an der Stelle, an der sie sich in einer Zelle befinden, an das Polymer geklebt, dann wird die Zelle selbst zerstört und - unter Zugabe von Wasser - wird das Polymer ausgedehnt und die strukturellen Details - nun etwa fünfmal größer - werden sichtbar.
Jede Technik für sich ist ein leistungsfähiges Mittel, um mehr über Systeme zu erfahren, die viel kleiner sind als das, was man normalerweise unter dem Mikroskop sieht, aber die Kombination der beiden Techniken ermöglicht es, noch tiefer in kleine Strukturen einzutauchen, die mit einer der beiden Techniken allein nicht aufgelöst werden könnten.
In unserem neuen Artikel zeigen wir, wie Einzelmolekül- und Expansionsmikroskopie kombiniert werden können, um einige der selteneren Proteine in Spalthefe zu untersuchen - ein SExY Protokoll. Dazu mussten wir eine Reihe von Hindernissen überwinden. So stellte sich zum Beispiel heraus, dass unsere Hefezellen sehr widerstandsfähige Zellwand-"Gürtel" haben, die der Verdauung widerstehen und ihre Ausdehnung einschränken, was zu sanduhrförmigen Zellen führt - SExY vielleicht, aber aus wissenschaftlicher Sicht nicht gerade nützlich! Außerdem lässt die Herstellung des Polymers und die Zerstörung der Zelle, um die Expansion zu ermöglichen, die fluoreszierenden Proteine nicht unversehrt, so dass wir verschiedene Aspekte des Protokolls optimieren mussten, um die Überlebensrate, die so genannte Retentionsausbeute der Fluorophore, auf ca. 50 % zu steigern. Zu den bemerkenswerten Neuerungen gehört die Zugabe eines Radikalvernichters bei der Herstellung des Polymers, wodurch die Belastung durch reaktive Chemikalien in diesem Schritt, die unsere fluoreszierenden Reporter schädigten, verringert wurde. In seiner optimierten Form kann das Protokoll nun verwendet werden, um Proteinstrukturen auf der Nanostrukturebene sichtbar zu machen - selbst für Proteine, die in geringer Häufigkeit vorkommen, wie die Kernproteine cbp1 und mis16 und das Zentromer-spezifische Histonprotein cnp1.
Das Projekt wurde 2016 in Marburg begonnen, wanderte mit den Autoren nach Pittsburgh und Paris und wurde schließlich hier in Bonn am IfMB fertiggestellt. Wir freuen uns, dass es nun im Open Biology Journal der Royal Society veröffentlicht wurde.